XLD Agar in der Stuhldiagnostik leicht erklärt
Der XLD (Xylose-Lysin-Desoxycholat) Agar gehört zum Standardrepertoire in der Stuhldiagnostik.
- Einführung:Mikrobiologie leicht erklärt: Nährmedien, ihre Typen und Anwendungen
- Blutagar einfach erklärt – Aufbau, Hämolyse und Anwendung
- MacConkey Agar erklärt: Selektiv- und Differentialmedium für gramnegative Bakterien
- Kochblutagar einfach erklärt: Optimalmedien für anspruchsvolle Erreger
- CLED Agar in der Urindiagnostik einfach erklärt
- XLD Agar in der Stuhldiagnostik leicht erklärt
Der XLD (Xylose-Lysin-Desoxycholat) Agar wird in der Stuhldiagnostik vor allem zur Differenzierung von Salmonellen und Shigellen gegenüber anderen Enterobakterien eingesetzt. Aufgrund dieser wichtigen Funktion gehört er, meist in Kombination mit weiteren Nährmedien, zur Standardausstattung vieler mikrobiologischer Labore.

Wer sich vorab einen Überblick über die verschiedenen Nährmedien verschaffen möchte, findet in meinem Artikel „Mikrobiologie leicht erklärt: Nährmedien, ihre Typen und Anwendungen“ eine kompakte Einführung.
XLD (Xylose-Lysin-Desoxycholat)-Agar
Die Zusammensetzung von XLD Agar unterscheidet sich geringfügig zwischen den Herstellern.
Zusammensetzung
- Xylose
- L-Lysin
- Laktose
- Saccharose
- Natriumchlorid
- Hefeextrakt
- Phenolrot
- Natriumdesoxycholat
- Natriumthiosulfat
- Ammoniumeisen(III)-citrat
- Agar Agar
- pH Wert 7,4
Der XLD Agar gehört gleichzeitig zu den Selektiv- und Differentialmedien. Was das bedeutet, schauen wir uns gleich genauer an.
XLD Agar als Selektivmedium
In der Stuhldiagnostik liegt der Fokus vor allem auf dem Nachweis gramnegativer Stäbchenbakterien. Um diese gezielt zu untersuchen, soll das Wachstum grampositiver Erreger unterdrückt werden. Im XLD-Agar übernimmt diese selektive Wirkung das Natriumdesoxycholat. Dabei handelt es sich um ein Salz der Desoxycholsäure, das einen Zellaufschluss sowie die Solubilisierung von Membranproteinen bewirkt und dadurch das Wachstum grampositiver Bakterien effektiv hemmt.
XLD Agar als Differentialmedium
Beim XLD-Agar wird es im Vergleich zu manch anderen Nährmedien etwas komplexer. Seine besondere Stärke liegt in der Kombination mehrerer Komponenten, die gezielt an den Differenzierungsprozessen beteiligt sind. Dazu zählen Xylose, Lactose und Saccharose, die Aminosäure L-Lysin sowie die Indikatorsubstanzen Phenolrot, Natriumthiosulfat und Ammoniumeisen(III)-citrat. Durch das Zusammenspiel dieser Bestandteile lassen sich verschiedene bakterielle Stoffwechseleigenschaften sichtbar machen und somit unterschiedliche Erreger voneinander unterscheiden.
pH-Indikator Phenolrot
Der pH-Indikator Phenolrot verleiht dem XLD-Agar seinen blass rötlichen Grundfarbton. Der pH-Wert des unbeimpften Agars liegt bei etwa 7,4 und damit im neutralen Bereich.
Kommt es durch bakterielle Stoffwechselprozesse zu einer Absenkung des pH-Werts, zeigt Phenolrot einen Farbumschlag nach Gelb. Dieser tritt in der Regel ab einem pH-Wert von etwa 6,6 oder darunter auf und weist auf ein saures Milieu hin. Steigt der pH-Wert hingegen in den basischen Bereich, färbt sich der Agar pink bis kräftig rot. Dieser Umschlag erfolgt ungefähr ab einem pH-Wert von 7,7. Durch diese pH-abhängigen Farbveränderungen lassen sich Säure- und Basenbildung im Rahmen des bakteriellen Stoffwechsels gut sichtbar machen.
Xylose, Laktose und Saccharose
Im zweiten Schritt richtet sich der Blick auf die im XLD-Agar enthaltenen Zuckerkomponenten, die eine zentrale Rolle bei der Differenzierung der Bakterien spielen.
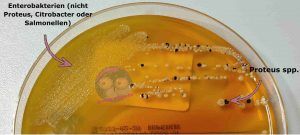
Die meisten Enterobakterien sind in der Lage Xylose, Laktose und Saccharose zu verwerten. Dabei entstehen unter anderem Essig-, Milch- oder Ameisensäure, was zu einer Absenkung des pH-Werts führt. Infolgedessen verfärbt sich der Agar gelb, und die Bakterien wachsen in gelben bis orangenen Kolonien. Enterobakterien wie Escherichia coli, Klebsiella spp., Enterobacter spp., aber auch Proteus spp. zeigen dieses Wachstumsmuster. Bei Proteus spp. besteht jedoch eine Besonderheit, auf die im weiteren Verlauf noch eingegehen werden.
Shigella spp. hingegen verstoffwechseln diese drei Zucker nicht. Der pH-Wert des Mediums bleibt daher weitgehend unverändert, sodass die Kolonien in einem zart rosafarbenen Ton erscheinen. Eine Ausnahme bildet Shigella sonnei, die Laktose sehr langsam fermentieren kann, in der Regel aber erst nach mehr als 24 Stunden Inkubationszeit.
Salmonella spp. wiederum kann Xylose abbauen aber die anderen Zucker nicht und wird dadurch zunächst ebenfalls eine Gelbfärbung des Mediums verursachen. An diesem Punkt kommt jedoch eine weitere Komponente des XLD Agars ins Spiel: L-Lysin.
L-Lysin
Einige Enterobakterien besitzen das Enzym Lysindecarboxylase, mit dem sie die Aminosäure L-Lysin zu Cadaverin decarboxylieren können. Zugegeben das klingt zunächst recht kompliziert. Schauen wir uns daher an, was dahintersteckt. Decarboxylierung bedeutet in diesem Zusammenhang, dass von der Aminosäure Lysin die Carboxylgruppe (–COOH) abgespalten wird. Diese wird in CO2 umgewandelt und freigesetzt. Als Reaktionsprodukt entsteht Cadaverin.
Cadaverin ist ein sogenanntes Diamin, also eine chemische Verbindung mit zwei Aminogruppen (–NH2), und besitzt einen basischen Charakter. Unter anderem Salmonella spp. verfügt über das Enzym Lysin. Durch die Bildung von Cadaverin wird die zuvor durch die Xylosefermentation entstandene Säure neutralisiert. Infolgedessen verfärben sich die Kolonien nicht gelb oder orange, sondern erscheinen rötlich bis pink.
Um zu verhindern, dass es auch bei anderen Enterobakterien mit Lysindecarboxylase-Aktivität zu einer Neutralisation kommt, sind Laktose und Saccharose im Überschuss im XLD-Agar enthalten. Deren Verstoffwechslung führt zu einer ausgeprägten Säurebildung, sodass der Agar trotz Lysinabbau gelb bleibt.
Natriumthiosulfat/Ammoniumeisen(III)-citrat
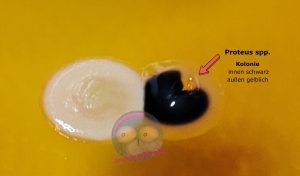
Bakterien wie Salmonella spp., Citrobacter spp. oder Proteus spp. sind in der Lage, Natriumthiosulfat zu Schwefelwasserstoff (H2S) zu reduzieren. Der freigesetzte Schwefelwasserstoff reagiert anschließend mit dem im Medium enthaltenen Ammoniumeisen(III)-citrat zu Eisensulfid (FeS), was zu einer schwarzen Verfärbung der Bakterienkolonien führt. Demnach können neben Salmonella spp. auch Proteus spp. und Citrobacter spp. schwarze Kolonien auf XLD-Agar ausbilden. Der entscheidende Unterschied liegt jedoch in der Färbung des umgebenden Agars sowie im Kolonierand.
Während Salmonella spp. typischerweise auf einem rötlichen bis pinken Agar mit schwarzem Zentrum wachsen, zeigen Proteus spp. und Citrobacter spp. häufig eine gelbliche Verfärbung des Agars, insbesondere im Randbereich der Kolonien. Diese Kombination aus H2S-Bildung, Koloniefarbe und Umgebungsfärbung des Mediums ermöglicht eine zuverlässige Differenzierung der relevanten Erreger.
Leider habe ich keine schönen Aufnahmen von einer Shigella oder Salmonella auf XLD Agar. Aber bei dem nachstehenden Link werdet ihr fündig.
Wachstum im Überblick
- Citrobacter gelb, opak, ev. schwarzes Zentrum und klare Ränder
- E. coli, Serratia, Enterobacter gelb, opak, ev. gelbes Präzipitat um die Kolonien
- Klebsiella spp. groß, gelb, hell, mucoid, opak, gelbes Präzipitat um die Kolonien
- Proteus mirabilis und P. vulgaris gelb, transparent, schwarzes Zentrum und klare Ränder
- Salmonella rot, transparent, schwarzes Zentrum
- Shigella rot, transparent
Fazit
Der XLD-Agar ist ein häufig eingesetztes Selektiv- und Differentialmedium in der Stuhldiagnostik. Durch die Kombination aus Zuckerfermentation, pH-Umschlag, Lysin-Decarboxylierung und H2S-Bildung lassen sich erste Hinweise auf unterschiedliche Enterobakterien gewinnen, insbesondere auf Salmonella und Shigella. Dabei ist der XLD-Agar nicht das einzige verfügbare Medium. Auch Hektoen-Agar oder SS-Agar werden als Alternativen oder ergänzend eingesetzt, je nach Laborroutine und diagnostischer Fragestellung.
Wichtig ist jedoch, dass die Ergebnisse auf XLD-Agar immer nur als Vorauswahl zu verstehen sind. Verdächtige Kolonien müssen anschließend durch weitere Verfahren wie MALDI-TOF-MS, zusätzliche biochemische Tests oder bei Salmonellen durch eine Serotypisierung abgesichert werden. Der XLD-Agar bildet damit einen sinnvollen ersten Schritt in der Diagnostik, ersetzt jedoch nicht die weiterführende Identifizierung.
Mehr über den Umgang mit gramnegativen Erregern und deren zunehmende Resistenz findest du in meinem Artikel „Antibiotikaresistenzen„.
Und wenn du noch mehr Lust auf Mikrobiologie bekommen hast, dann schau dir doch mal mein tolles Quizbuch an.
Quellen